Zeig‘ mir deinen Hirnscan und ich sag‘ dir, wie alt du wirklich bist
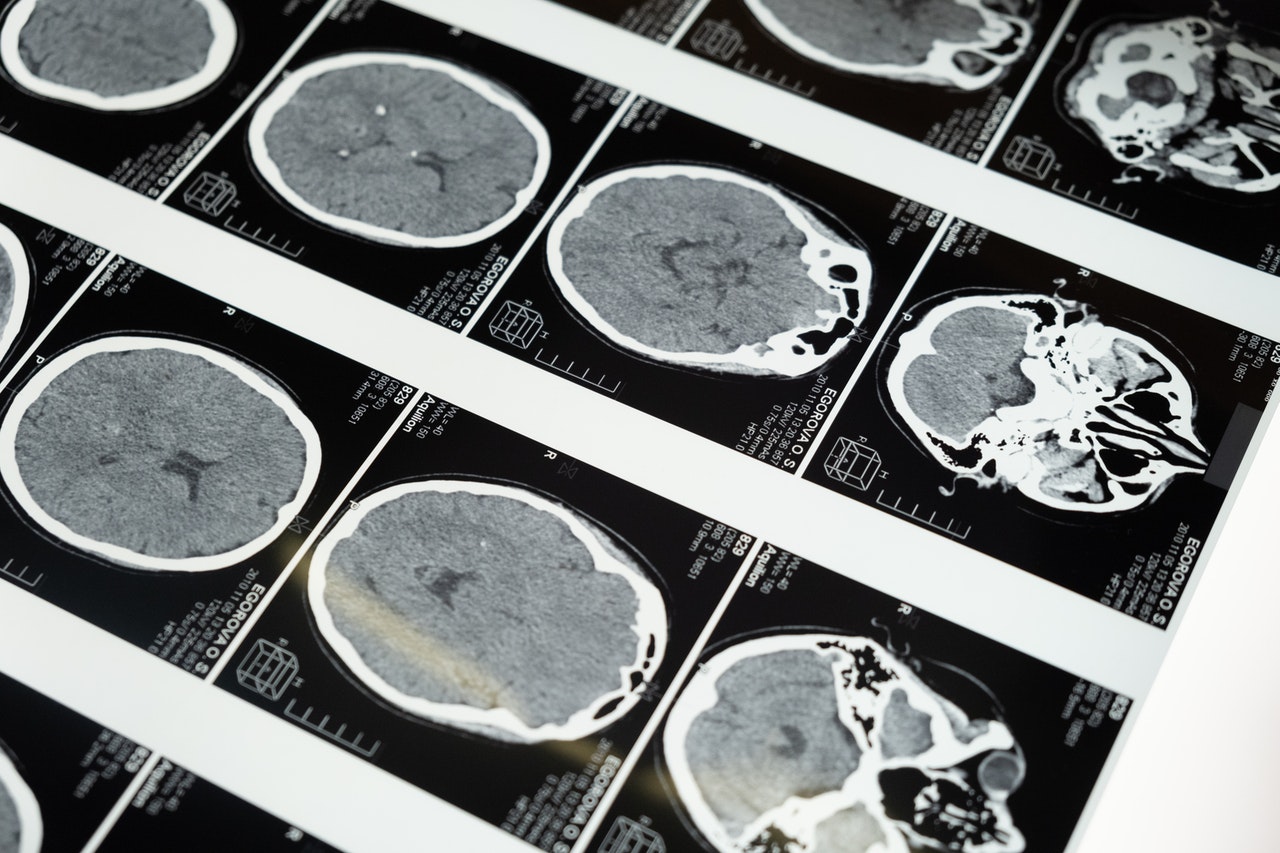
Aus Hirnbildern lässt sich mithilfe sogenannter Künstlicher Neuronaler Netzwerke das biologische Alter eines Menschen genau bestimmen. Bislang war jedoch unklar, anhand welcher Merkmale die Netzwerke auf das Alter schließen. ForscherInnen des Max-Planck-Instituts für Kognitions- und Neurowissenschaften haben jetzt einen Algorithmus entwickelt, durch den sich zeigt: Die Altersschätzung geht auf eine Bandbreite an Merkmalen im Gehirn zurück, und gibt dabei generelle Auskunft über den Gesundheitszustand eines Menschen. Der Algorithmus könnten damit helfen, Tumore oder Alzheimer schneller zu erkennen und erlaubt Rückschlüsse über die neurologischen Konsequenzen von Erkrankungen wie Diabetes.
Tiefe Neuronale Netzwerke sind eine KI-Technologie die unseren Alltag schon heute auf vielen Ebenen bereichert: Die künstlichen Netzwerke, die der Funktionsweise echter Neuronen nachempfunden sind, können Sprache verstehen und übersetzen, Texte interpretieren und Objekte und Menschen in Bildern erkennen. Sie können aber auch das Alter einer Person anhand eines MRT-Scans ihres Gehirns bestimmen. Zwar ließe sich das Alter auch einfacher herausfinden, indem man die Person danach fragt. Anhand der maschinellen Altersbestimmung bekommt man jedoch auch eine Vorstellung davon, wie ein gesundes Gehirn in verschiedenen Lebensphasen im Normalfall aussieht. Schätzt das Netzwerk das biologische Alter des Hirns anhand des Scans höher ein als es tatsächlich ist, kann das auf mögliche Erkrankungen oder Verletzungen hinweisen. Frühere Studien hatten etwa herausgefunden, dass die Gehirne von Menschen mit bestimmten Erkrankungen wie Diabetes oder starken kognitiven Einschränkungen scheinbar mehr Jahre auf dem Buckel hatten als es eigentlich der Fall war. Die Gehirne waren also in einem biologisch schlechteren Zustand als man es aufgrund des Alters dieser Menschen annehmen würde.
Obwohl die Künstlichen Neuronalen Netzwerke das biologische Alter präzise bestimmen können, wusste man bisher nicht, welche Informationen aus den Gehirnbildern ihre Algorithmen dafür nutzen. Wissenschaftler aus der KI-Forschung sprechen hier auch vom „Black Box Problem“: Demnach schiebt man ein Hirnbild in das Modell, die „Black Box“, lässt es von diesem verarbeiten – und bekommt letztendlich nur dessen Antwort. Wie diese Antwort zustande kommt, war jedoch aufgrund der Komplexität der Netzwerke bislang unklar. Wissenschaftler des Max-Planck-Instituts für Kognitions- und Neurowissenschaften (MPI CBS) in Leipzig wollten daher die Black Box öffnen: Worauf schaut das Modell, um zu seinem Ergebnis, dem Hirnalter, zu gelangen? Dafür entwickelten sie gemeinsam mit dem Fraunhofer-Institut für Nachrichtentechnik in Berlin einen neuen Interpretationsalgorithmus, mit dem sich die Altersschätzungen der Netzwerke analysieren lassen.
„Wir haben zum ersten Mal den Interpretationsalgorithmus in einer komplexen Regressionsaufgabe angewendet“, erklärt Simon M. Hofmann, Doktorand am MPI CBS und Erstautor der zugrundeliegenden Studie, die jetzt im Fachmagazin NeuroImage erschienen ist. „Wir können nun genau bestimmen, welche Regionen und Merkmale des Gehirns für ein höheres oder niedrigeres biologisches Alter sprechen.“
Dabei zeigte sich: Die Künstlichen Neuronalen Netzwerke nutzen unter anderem die weiße Substanz, um Vorhersagen zu treffen. Sie schauen demnach insbesondere darauf, wie viele kleine Risse und Vernarbungen sich durch das Nervengewebe im Gehirn ziehen. Zudem analysieren sie, wie breit die Furchen in der Großhirnrinde, dem Cortex, sind oder wie groß die Hohlräume, die sogenannten Ventrikel. Aus früheren Studien weiß man, je älter eine Person ist, desto größer sind bei ihr im Schnitt die Furchen und Ventrikel. Das Interessante dabei: Die Künstlichen Neuronalen Netzwerke waren selbstständig zu diesen Ergebnissen gekommen – ohne dass man ihnen diese Information gegeben hatte. Während ihrer Trainingsphase standen ihnen lediglich die Hirnscans und die wahren Lebensjahre der Person zur Verfügung.
„Natürlich kann man eine erhöhte Altersschätzung auch als Fehler des Modells interpretieren,“ so die Leiterin der Forschungsgruppe, Veronica Witte. „Wir konnten aber zeigen, dass diese Abweichungen biologisch bedeutsam sind.“ So bestätigten die ForscherInnen zum Beispiel, dass Menschen mit Diabetes ein erhöhtes Hirnalter haben. Sie konnten zeigen, dass bei den Betroffenen mehr Läsionen in der weißen Substanz auftreten.
Schon heute ist klar, Künstliche Neuronale Netzwerke werden eine zunehmend wichtigere Rolle bei der medizinischen Diagnose einnehmen. Zu wissen, woran sich diese Algorithmen orientieren, wird damit immer wichtiger: In Zukunft könnte ein Hirnscan von verschiedenen Netzwerken automatisch analysiert werden, die sich jeweils auf bestimmte Bereiche spezialisiert haben – eines zieht Rückschlüsse auf Alzheimer-Erkrankungen, das andere auf Tumore, und wieder ein anderes auf mögliche psychische Störungen. „Die Medizinerin bekommt dann nicht nur Rückmeldungen, dass womöglich bestimmte Erkrankungen vorliegen. Sie sieht auch, welche Bereiche im Gehirn den Diagnosen zugrunde liegen“, erklärt Hofmann. Die entsprechenden Merkmale werden durch die Algorithmen jeweils direkt im MRT-Bild markiert und können so leichter von den MedizinerInnen entdeckt werden – diese wiederum können dann unmittelbar Rückschlüsse daraus ziehen, wie schwer eine Erkrankung ist. Zudem ließen sich Fehldiagnosen leichter entdecken: Wenn die Analyse auf biologisch unplausiblen Bereichen basiert, etwa auf Fehlern, die beim Erstellen des Bildes entstanden sind, können diese unmittelbar von der Ärztin erkannt werden. Der Interpretationsalgorithmus des Forschungsteams kann damit letztendlich auch dazu beitragen, die Genauigkeit der Künstlichen Neuronalen Netzwerke selbst zu verbessern.
In einer Folgestudie wollen die ForscherInnen nun genauer untersuchen, warum ihre Modelle auch auf Merkmale im Gehirn schauen, die bislang in der Altersforschung kaum eine Rolle spielten. Es hatte sich beispielsweise herausgestellt, dass die Neuronalen Netzwerke sich auch auf das Kleinhirn fokussieren. Wie Alterungsprozesse in gesunden und erkrankten Menschen dort voranschreiten, ist WissenschaftlerInnen bislang ein Rätsel.
Originalpublikation:
Simon M Hofmann, Frauke Beyer, Sebastian Lapuschkin, Ole Goltermann, Markus Loeffler, Klaus-Robert Müller, Arno Villringer, Wojciech Samek, A Veronica Witte. Towards the interpretability of deep learning models for multi-modal neuroimaging: Finding structural changes of the ageing brain. Neuroimage. 2022 Nov 1;261:119504. doi: 10.1016/j.neuroimage.2022.119504. Epub 2022 Jul 23.
Foto: Pexels/ cottonbro